먼저 굳이 인텔맥 Big Sur일 필요는 없다는 점을 밝혀둡니다. 다만, 저는 설정을 할 당시 그 시점의 환경을 기입해두곤 합니다. 이는 제가 모든 환경을 테스트 해 볼수도 없는 노릇이고, 그냥 쓰던 환경을 다시 쓸수있게 해놓고는 간단한 후기를 남기는 정도입니다.
최근 시스템 설정을 마치고 논문작업에 바쁜 나날을 보내다가 잠깐 여유를 부려 작성중입니다. 이는 지난 번 글(1, 2)에서 밝혔다시피 새로운 시스템을 설정하는 김에 그 동안 미뤄둔 환경설정들을 모두 업데이트하면서 작성하는 글입니다. 현재 작성해둔 글의 경우에는 다음 두 글이 있습니다.
– Intel oneAPI (Intel compiler / MKL) 환경설정
본격적인 설정 글들이 끝나고 나면, 작업 워크플로우에 대한 기록을 남겨볼 예정입니다. 본격적인 작업 환경에 대한 이야기 역시 몇 단계로 나누어서, 기본적인 도구들을 이용해 어떻게 데이터를 구성하고, 이를 분석하는가에 대한 이야기 뿐만 아니라, 필자의 논문작성 환경에 대해 소개하면서 각 단계가 어떻게 구성되어있는지에 대해 정리를 해 볼 예정입니다.
필자는 연구분야 종사자로서, 일반적인 개발자 분들에 비해 전문성이 부족할수 있음을 미리 밝혀두는 바입니다.
0. 들어가기 앞서
앞선 글에서 말씀드렸다시피, 필자의 맥북에서는 여러가지 개발환경이 혼재되어 있습니다. 이 자체가 디폴트로서, 센터 전반적으로 Mac등의 시스템 관리팀에서 편의스크립트등을 제공하면서 환경변수를 선택할 수 있게 해주곤 하는데, 여기서는 기본적으로 설치된 Python 3.7/3.9 버전을 기반으로 진행하도록 하겠습니다.
중요한 것은, 아직까지 Python 2.7버전이 과학분야에서 완전히 도태되지 않았습니다. 상당히 많은 옛날 스크립트 (제가 예전에 만든것 포함)이 레거시로 남아있는 경우가 많기때문에, 2.7환경 역시 필요시 사용할 수 있어야 합니다. 여기서 중요한 것이, 사실 virtualenv를 사용하면 매우 깔끔하게 관리되곤 하는데, 시스템 전체의 기본 환경을 개인 사용자환경에 맞추다보니 왠만하면 프로젝트별로 따로 구분하지 않으려고 합니다. 이는, 개발자 분들과 달리 제가 쓰는 코드의 주 사용자는 저이고, 논문 투고하면서 오픈소스로 일부를 업로드 하곤 합니다. 이렇게 업로드한 소스코드의 경우에도 일반적으로 과학자들이 많이 사용하는 numpy/scipy/matplotlib를 기반으로 진행하게 되므로 특별히 다른 환경설정을 해 주는 것이 오히려 번거로운 경우가 많이 있습니다.
Python 개발환경에서 (특히 과학자들에게) Mac에서 흔히 쓰이는 패키지 관리소는 brew뿐만 아니라 pip, anaconda등이 있습니다. 상당히 많은 과학자분들이 anaconda환경을 사용하시고, 이 속에는 다양한 연구용 도구들이 함께 설치되어있는 장점이 있습니다. 다만 필자의 경우에는, 기존의 관성에 인해 brew/pip/conda를 모두 혼용하곤 했었는데, 가끔 문제도 생기고 어느순간 문제를 추적하기가 매우 힘들어진 것을 깨닫고는 분리해서 사용하고 있습니다. Anaconda의 경우에는 사용하고 있는 맥북에서 이미 설치되어있습니다만, 제가 사용하지는 않습니다. 그렇다고 모두 날려버리기에는, /usr/local 폴더가 주기적으로 하는 업데이트를 통해서 서버에서 통채로 받아와서 덮어씌우기 때문에 개인화 설정을 마구잡이라 할 수 없는 단점이 이 있으므로, 그냥 무시하고 있습니다. Python패키지는 pip로, 그 외 대부분의 도구들은 brew로 진행합니다. 그러므로 오늘 이야기는 기본적으로 pip를 이용한 설치 및 사용을 배경으로 합니다. 중요한것은 pip역시 기반 Python버전에 따라 달라지므로 python -m pip를 기반으로 진행합니다.
Python기본 코드 뿐만 아니라 Jupyter환경 역시 Python을 활용하시는 과학자분들에게 익숙할 것이라 생각됩니다. 현재 Python자체 점유율보다 Jupyter의 점유율이 압도적으로 높은 걸로 알고 있습니다. 물론 R이나 Julia도 포함된 숫자이긴 합니다만… 저의 경우 Jupyter notebook확장자인 ipynb를 주된 작업 폴더 (저는 대부분 제가 직접적으로 하고 있는 일들을 다루는 폴더)에서 “find . -name “*.ipynb” | wc -l” 로 카운팅해보면 약 400개가 나옵니다. 여기에는 git branch등과 관련된 허수가 끼여져 있으나, 대략적으로 200여개는 된다고 볼 수 있습니다. 같은 방법으로 해보면 총 .py의 카운터가 1500여개, .sh의 카운터가 얼추 450개입니다. “.cpp”는 120여개 정도로 나오네요. 즉 jupyter notebook 파일이 .sh의 개수만큼 있을정도로 많은 활용을 하고 있다는 이야기입니다.
1. Python
1-1. 기본 패키지 설치
이미 Python은 설치되어있다고 보고, 기본 python에 필요한 alias를 추가해줍니다. 현재는 디폴트 python커맨드에 제가 사용하는 버전이 할당되어있어서 건드리지 않았습니다. 먼저 과학자에게 필수적인 세개의 패키지를 설치합니다.
python -m pip install numpy scipy matplotlib
Numpy의 경우 numerical python 패키지로서 각종 기본 도구들이 들어있습니다. 예로들어 기본 array에 비해 Numpy의 array는 Matlab과 마찬가지로 행렬 계산을 편하게 진행해줍니다. 이외에 자세한 도구가 많이 있는데, 필자의 경우에는 numpy만큼은 “from numpy import *” 로 다른 이름을 부여하지 않고 가져오는 편입니다.
Scipy의 경우 Scientific Python 패키지로서 과학에서 흔히 쓰이는 여러 표준 수치해석 코드들이 모여 있습니다. 많은 코드의 경우 일종의 래퍼를 제공하는 형태이기때문에, Scipy에서 제공되는 함수들의 경우 빠르고 안정적으로 작동합니다. OpenBLAS등과 같은 다른 오픈소스의 래퍼 함수들을 제공하는 경우가 많이 있으므로 Python으로 연구를 진행할때 꼭 불러오게 되는 도구입니다. C++에서 MKL이나 GSL에 해당되는 요소라 생각해도 무방합니다.
Matplotlib의 경우 기본적으로 Matlab의 plot기능과 유사하게 제공되는데, 필자의 경우 현재까지 대부분의 연구결과 그래프를 해당 패키지를 이용해서 그렸습니다. 기본 플롯의 경우에는 Gnuplot과 마찬가지로 간편하게 이용할 수 있는데, 연구용 그래프 포맷은 사실 그때그때 고려할 부분이 많고 해서 대부분의 경우 구글검색 혹은 과거에 직접 사용한 갤러리에서 선택하는 편입니다. 일반적으로 학회/세미나용 그래프들은 아래 소개할 Jupyter정도에서 만들어 두는 편입니다만, 논문용 그래프의 경우에는 Matplotlib를 기반으로 각 Figure당 한개의 스크립트를 대응시켜서 만들어 둡니다. 이는, 일반적으로 논문 작업의 경우 폴더가 통채로 git을 통해 추적되고 있기 때문에, 어느 시점에서 해당 데이터 및 그래프를 재가공하기 수월하기 때문입니다. 또한, 기본 스타일 스크립트를 미리 구성해서 각각의 figure용 코드에서 기본 스타일을 로드해오기만 하면, 전체 그래프 스타일을 한번에 바꾸기 용이합니다. 가끔 제법 시간이 걸리는 후처리 가공이 포함될때도 있는데 – 개인적으로는 최종 그래프용 스크립트에서 후처리 가공을 빼려고 노력합니다만, parameter estimation등이 포함되는 경우에는 함께 진행하는게 더 편리한 경우가 많이 있습니다 – 일반적으로는 GNU’s parallel명령어로 전체 플롯을 재수행하곤 합니다. 각각의 .py에는 이미 Tex파일에서 include시키는 각 파일명과 참조폴더를 이미 설정해두었기 때문에, 다음과 같이 전체 플롯을 업데이트 시키고, Tex파일을 재컴파일 하는것으로 그래프만 업데이트 시키는 편입니다.
parallel python {} ::: $(ls plot_fig*.py)
1-2. Parallel computing
단독으로 사용가능한 여러 코드를 한번에 시행하려면 GNU’s parallel 커맨드가 좋습니다. 그런데 가끔은, Python내에서 참조하는 계산들에서 병렬화가 필요할 때가 있습니다. 실제로 고성능 계산이 필요한 경우에는 C++를 사용한다고 했지만, 꼭 그렇지 않더라도 따른 개발 시간을 가져가지 않고 현재 코드에서 조금의 수정으로 몇배정도 빠르게 결과를 나오길 기대할 수 가 있지요. 그럴 경우에는 Multiprocessing 패키지를 이용하면 됩니다. 다만, 이걸 쓰고 나면 나중에 소개할 기본 cProfile 명령어로 분석이 힘들어지다보니 막상 필요할때만 쓰는것을 추천합니다. 설치는 간편합니다.
Python -m pip install multiprocess
일반적으로, 데이터 처리에 쓰이는 경우 행렬로 주어진 데이터들을 각 요소별로 나누어 처리하는 경우가 많기에 다음과 같은 레퍼런스 하나 두고 그때그때 복사해서 쓰는 편입니다. 다음번에 보면 기억안나요. 다음 예시는 user_fcn을 정의해두었고, arg_list와 같은 arguments를 가져온다고 본 예시입니다.
import multiprocessing as mp N_PROCESSES = 8 pool = mp.Pool(N_PROCESSES) arg_list = [(A_arr[i], B_arr[i], C_arr[i], D_const, E_const) for i in range(N_dat)] re = pool.starmap(user_fcn, args_list) pool.close() pool.join()
이와는 별개로 scipy등에서 사용하는 패키지등이 자체적으로 병렬화가 되어있을수 있습니다. 대표적으로 OpenBLAS같은 경우인데, 이경우 디폴트 설정이 저같은 경우 모든 코어를 사용하는 경우로 나옵니다. 다만, 저의 경험에서는 Python에서 계산시키는 경우 그 목적 및 문제의 규모 자체가 C++의 계산 코드와 다르다보니 모든 코어를 사용한 연산이 크게 중요하지 않은 경우가 많이 있습니다. 이 경우에는 해당 환경변수를 터미널에서 export시켜주어야 합니다. 예를들어 OpenBLAS의 경우 다음과 같습니다. 숫자는 원하는 쓰레드 숫자를 넣어주면 됩니다.
export OPENBLAS_NUM_THREAD=8
1-3. Python 사용 스타일 및 Emacs 관련 환경설정
덧붙여, Emacs의 Python모드는 매우 편리한 도구입니다. 디폴트로는 C-c C-p가 M-x run-python으로 매핑되어 있습니다. Python shell interpreter로서 매우 생산적입니다. 다만 기본 설정이 현재 원하는 python interpreter이라는 보장이 없으므로 다음과 같이 .emacs에 설정해줄 필요가 있습니다. 원하신다면 사용하시던 인터프리터를 확인하셔서 입력하시면 됩니다 (e.g., which python).
(setq python-shell-interpreter "/usr/local/bin/python3")
필자의 경우 Jupyter를 제외하면 단일 목적 코드이든 (터미널에서 Emacs), 조금 긴 이론계산등의 코드이든 (Emacs GUI) Emacs에서 Python 작업을 진행합니다. 이 경우 ‘기본적인 방침’은 각 함수에 doc string을 만들어 주고 arguments/returns에 대한 값과 단위를 기입해둡니다. dimensionless variable역시 기본 unit을 기입해두곤 합니다. 만약, publish된 내용을 기반으로 한 코드인 경우 해당 수식 번호를 넣어두기도 합니다. 그리고 main함수 구성에서 (__name__==’__main__’) 각 함수의 doc string을 출력시켜 줍니다. 이로인해 터미널에서도 혹은 Emacs 환경에서도 즉각적으로 필요한 함수들을 찾을 수 있곤 합니다. 많아지면, doc string을 그대로 출력하기보다는 기본 함수명정도만 이야기하는 경우도 있습니다.
def f1(a, b, x): """ Test f1: This will calculate ... Arguments: a (unit): meaning a b (unit): meaning b x (unit): meaning L Returns: value = a + b + x """ return a+b+x def f2(t, p, q): """ Test f2: This will calculate ... Arguments: t (unit): meaning t p (unit): meaning p q (unit): meaning q Returns: value = t*p*q """ return t*p*q if __name__=='__main__': print(f1.__doc__) print(f2.__doc__)
1-4. cProfile
Single thread기반의 기본적인 코드라고 하더라도 Python자체가 제법 느린편이다보니, 미적분 및 self-consistency등 이공계의 수치해석의 기본적인 방법론들은 제법 무거운 nested loop를 계산하게 됩니다. 저에게 기본적으로 중요한 순서들은 (a) 일단 만든다, (b) 정확한지 확인한다, (c) 코드의 안정성 및 최적화를 진행시킨다 라고 생각합니다. 어찌보면 당연한데, 의외로 (c)의 경우 많은 과학자분들이 스킵하기도 합니다. 저 역시, Python으로 구성하는 대부분의 코드들은 (1) 그냥 아이디어 스크래치용이거나 (2) 성능이 별로 중요하지 않거나 한 경우입니다. 그래서 둘다 위의 (c)단계를 스킵하는 경우가 제법 자주 있습니다.
보통 실행해서, 1분이내의 코드들은 무시하는 편입니다만, 그래도 몇분정도 이상 걸리는 코드의 경우는 한번씩 time profile을 확인해주는 편입니다. 다양한 도구가 많이 있겠지만, 저에게는 기본적으로 사용가능한 cProfile이 좋습니다. 다만 multiprocessing의 경우에는 별 도움이 안되는 경우가 많이 있습니다. 만약 본인의 코드가 test.py고 기본 파싱 인풋이 input.dat라면 다음처럼 report.prof라는 프로파일 리포트 데이터를 만들 수 있습니다.
python -m cProfile -o report.prof test.py input.dat
여러 뷰어가 있겠지만, 필자의 경우에는 현재 snakeviz를 씁니다. 예전에는 다른거 썻는데, 특별히 의미를 주는 것 보다는 OS 업데이트 되다가 호환 잘되는거 검색해서 쓰는 편입니다.
python -m pip install snakeviz snakeviz report.prof
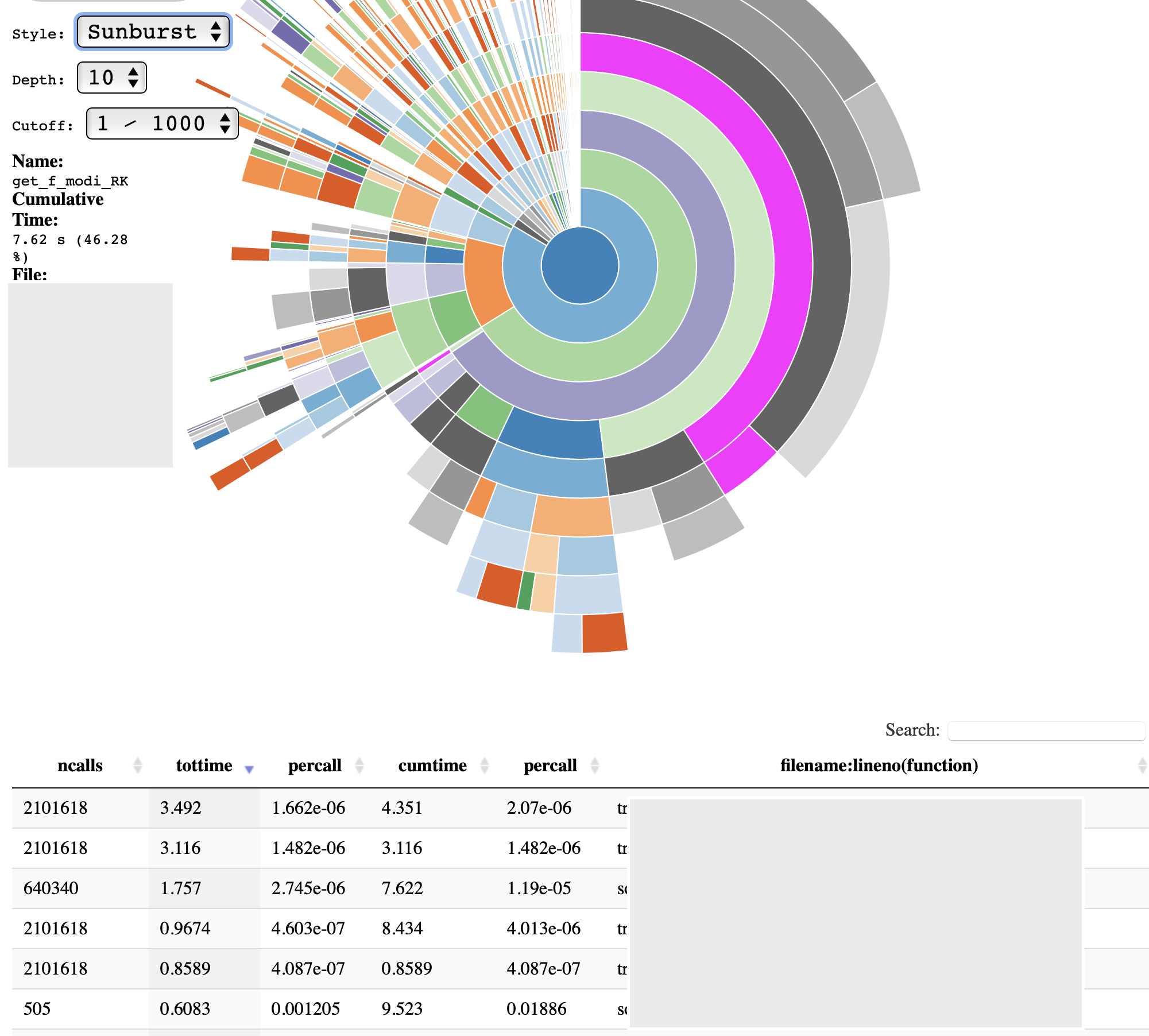
현재 제가 사용한 예시 코드의 경우 다음과 같은 결과를 볼 수 있습니다. 해당 코드의 원형은 사실 multiprocessing이 적용된것이지만, 저는 이 경우에도 single thread기반 코드를 남겨두는 편입니다. 이 경우 총 계산시간이 약 10분정도인데, 초반에 루프 몇개만 돌렸을때의 리포트입니다. get_f_modi_RK에서가 약 46.28

2. Jupyter notebook (or lab)
위에서 Python 개발 환경및 기본적인 사용철학에 대한 이야기를 간략하게 소개한 바 있습니다. 말씀드린 바와 같이, 필자의 경우 Python 코드 자체에 집중하는 경우에는 항상 Emacs환경에서만 사용합니다. 그게 터미널이던지 GUI던지 차이가 있겠지만요 (각기 용도가 다릅니다만, 어차피 경험에 따른 사용철학의 차이이므로 지금은 중요한 문제가 아닌 것 같습니다). 그런데, 그 외에 Python을 많이 사용하는 환경은 바로 Jupyter notebook입니다. 이 경우는 ‘연속적인’ 이론/데이터 분석이 중요한 경우이고, 사실상 연구의 대부분이 여기에 속한다고 생각합니다. Notebook환경은 매우 많은 연구용 플랫폼이 제공합니다. 필자가 애용하는 또 다른 중요한 노트북 환경은 Mathematica로서, 순수 이론계산을 하면서 symbolic calculation이 중요한 것은 (비록 Python의 sympy가 있더라도) Mathematica에서 이루어집니다. 이 경우에도 핵심은, 수치적인 계산 결과는 data로 export시킨다음, Python에서 처리합니다. 결과적으로 Python는 필자의 연구에서 핵심적인 데이터처리를 담당하고 있고, Jupyter notebook의 경우에는 다양한 환경에서 생성된 복잡한 데이터 인터페이스를 손쉽게 알수있게 구성하고 실수를 줄이기 좋게 되어있습니다. 예를들어 다음 스크린샷의 경우 Markdown cell에서 특정 data의 구조를 기입해둡니다. 이때 각 물리량의 unit까지 함께 기입합니다. 의외로 unit conversion에서 많은 실수가 일어나므로 개인적으로 매우 중요하다고 생각합니다.

전체적인 인터페이스는 다음 스크린샷과 같은 모습입니다. 해당 연구에서 사용하는 데이터는, 제가 짠 코드로 계산한것과 아닌게 섞여있습니다. 더더군다나, 다른사람이 짠 코드 역시 그 전의 사람이 짠 코드가 내는 결과부분과 또 다른사람이 추가한 부분의 결과부분을 함께써야 합니다. 제일 속시원한것은 제 스타일로 코드를 수정하는 것이라 생각합니다. 다만, 많은 경우 코드 개발/유지보수에 너무 많은 시간이 할애하게 되므로 결국 제 코드만 손보고는 다른사람의 코드의 경우 불편한 인터페이스라도 거기에 맞춰서 사용하는 경우가 많이 있습니다. 그러면 위의 데이터 처리 관련에서 기본 column에 해당되는 설명 및 base unit을 각기 기술해주고, 그런 부분들이 아래 contents에 보이는 각 subsection들마다 조금씩 다르게 정의되는 편입니다. 그리고 한번 이런 작업을 해두고 나면, Jupyter 내부에서는 저의 방식으로 데이터들이 재정리되고, unit conversion의 실수도 많이 줄어들기 마련입니다.

또한 매우 중요한 부분이 있는데 jupyter notebook의 경우에는 server-client의 개념으로 접속이 가능합니다. 다시말해 연구실에 있는 워크스테이션에서 노트북 서버를 열어두고 랩탑에서 접속해서 쓰기도 합니다. 과거 원통형 맥 프로를 사용할때는 필자도 많이 사용했던 기능이었습니다만, 요즘은 맥북프로만을 주력으로 사용하면서 잘 사용하지 않고 있습니다.
2-1. Jupyter의 설치 및 기본설정
매우 다양한 패키지 관리소들이 Jupyter를 지원합니다만, 필자의 경우에는 Python과 관련된 모든 패키지들을 pip로 통일시켜둡니다. 여기에 나중에 필요한 extensions들을 설치하기 위해 추가 도구들을 설치해주게 됩니다.
python -m pip install jupyter
설치후 jupyter notebook을 실행하면 되는데, 문제는 이 때 열리는 웹브라우저는 시스템 기본 브라우저입니다. 필자의 경우 사파리 / 크롬은 각각의 용도로 많이 사용하기에, 잘 사용하지 않는 파이어폭스를 Jupyter기본 브라우저를 설정하였습니다. 이렇게 잘 사용하지 않는 브라우저를 지정하면 현재 진행되고 있는 ‘중요한 연구’가 다른 참조 페이지들에게 파묻히는것을 방지할 수 있습니다. 또한, 한창 연구를 진행 중일때 약 4-5개의 노트북이 떠 있는 경우가 흔하기 때문에 관리상의 편의도 있습니다. 일단 새로운 설치인 경우 설정파일을 생성시키도록 하겠습니다.
jupyter notebook --generate-config
이제 ~/.jupyter 폴더가 생성되고 여기에 jupyter_notebook_config.py가 생성됩니다. 해당 파일을 열어서 다음 부분을 원하는 브라우저에 맞게 수정해주면 됩니다. 필자의 경우에는 앞서 말씀드렸다시피 파이어폭스를 지정해 두었습니다.
c.NotebookApp.browser = u'open -a /Applications/Firefox.app2-2. Jupyter notebook extensions
다른 개발도구들과 마찬가지로, Jupyter역시 다양한 도구를 활용할 필요가 있습니다. 보통 재설치할때마다 구글을 찾아보는 편인데, 현재는 다음 페이지를 참조하여 진행했습니다.
python -m pip install jupyter_contrib_nbextensions python -m pip install https://github.com/ipython-contrib/jupyter_contrib_nbextensions/tarball/master jupyter contrib nbextension install --user python -m pip install jupyter_nbextensions_configurator jupyter nbextensions_configurator enable --user이후 Jupyter notebook을 실행해보면 Nbextensions탭이 생기고 여기서 활성화시키는 도구들을 선택할 수 있습니다. 필자의 경우 늘 활성화 시키는 도구들은 (1) Table of Contents로서 위 화면에 보이는 Contents부분입니다. Markdown cell에서 #, ##, ###헤딩을 읽어들여서 자동으로 분류해주는 부분입니다. 이는 제법 연구분석 노트가 긴 경우 매우 유용합니다. 덧붙이자면 (2) Collapsible Headings를 함께 활용하면 매우 긴 연구노트에서 손쉽게 네비게이션을 할 수 있게 됩니다. 의외로 중요한 도구입니다. 여기에 (3) executed time을 추가해주곤 하는데, 가끔 외부 Python코드를 수행할때 도움이 됩니다.
기본 도구팩에 포함되지 않는 자원사용량에 해당되는 도구가 있습니다 (링크). 어차피 CPU자원의 경우 istat을 활용하고 있지만, 다양한 데이터들을 가져오는 입장에서는 해당 노트북의 메모리 사용량을 보여주는 기능은 매우 유용합니다. 해당패키지는 추가설치가 필요합니다.
python -m pip install jupyter-resource-usage2-3. 매직 커맨드 (
Jupyter의 경우 Ipython kernel을 활용합니다. 그래서 Ipython의 여러 다양한 그리고 강력한 매직 커맨드를 사용할 수 있습니다. 여러 매직 커맨드는 다음 메뉴얼에 잘 소개 되어있습니다. 이를 사용시 용도에 맞춰
마찬가지로 터미널 커맨드를 지원!합니다. 그래서 가끔 현재 폴더를 굳이 터미널에서 열어야 할 수고를 덜어줍니다. 예를들어 다음 커맨드들은 늘 사용하게 되더군요. 의미야 너무 뻔하니 설명을 덜도록 하겠습니다.
!cat input.dat | grep const_A !pwd !cp fig_a.pdf ../../manuscript/figs/. !open .
Leave a Reply